GenoScreen Expertise
GenoBiome® - Solution de diagnostic tout-en-un pour le microbiome
La génomique a révolutionné l'étude des microbiomes en permettant une analyse approfondie de l'ADN des micro-organismes présents. Basé sur cette approche, GenoBiome® permet de caractériser la diversité microbienne, d'identifier des espèces spécifiques et d'étudier les interactions entre ces micro-organismes et leur environnement.
Ces connaissances offrent de nouvelles opportunités dans le développement de produits respectant les communautés bactériennes symbiotiques dans différents domaines d'application, tels que :
Contactez-nous pour plus d'informations
Une solution intégrée pour l'étude des communautés microbiennes
GenoBiome® propose une gamme complète de services pour l'analyse des communautés microbiennes, avec des protocoles analytiques et bioinformatiques conçus spécifiquement pour la matrice étudiée (peau, sol, intestin).
L'objectif de cette solution est de vous fournir le support scientifique, l ' analyse technique et l' assistance adaptée nécessaires pour mener votre projet à une résolution complète de vos questions biologiques.
Nos services de conseil, assurés par des docteurs en écologie microbienne ou en génomique, favorisent une approche holistique de la recherche scientifique et mettent l'accent sur la répétabilité et la reproductibilité des résultats expérimentaux.
Découvrez des rapports interactifs adaptés à vos besoins, qui facilitent l'analyse approfondie et la compréhension de données complexes. En outre, vous bénéficiez d'une assistance complète pour l'établissement de rapports sur les données, ce qui améliore la transparence et la clarté de la communication scientifique.
Rapports interactifs
Les rapports fournis avec GenoBiome® peuvent être adaptés à l'objectif de votre projet :
Recherche
Un niveau élevé de détails scientifiques et le soutien d'experts en génomique
Marketing
Des résultats exploitables qui peuvent être utilisés pour formuler des allégations, avec un soutien pour leur utilisation
Réglementaire
Transparence et aide à la constitution des dossiers
Nos rapports bioinformatiques sont conçus pour fournir une expérience globale, vous aidant à comprendre vos résultats avec facilité.
- Rapport facile à parcourir : Zoomez, filtrez et explorez vos données directement dans le rapport, sans outils supplémentaires
- Visualisations enrichies : Graphiques et tableaux interactifs accompagnés d'explications claires
- Contexte complet : Obtenez une meilleure compréhension grâce à des annotations détaillées
- Méthodologie transparente : Accédez à une explication étape par étape des méthodes d'analyse utilisées

Laissez nos experts vous guider dans votre projet

Plan expérimental
Concevez votre projet avec des experts qualifiés en microbiome, à partir de votre question biologique.
Stratégie d'échantillonnage
Notre assistance s'étend à la mise en œuvre d'une stratégie d'échantillonnage standardisée pour la production de données fiables et l'interprétation précise des résultats. Nous offrons également un soutien pour la collecte d'échantillons avec :
 Kit d'échantillonnage
Kit d'échantillonnage
Un kit d'échantillonnage peut être fourni avec un protocole de conservation pour garantir le maintien de l'intégrité pendant le transport.

Analyse d'échantillons sur mesure
Notre large portefeuille technique permet de répondre aux besoins de vos projets, qu'ils soient basés sur le séquençage à lecture courte (short read), la technologie à lecture longue (long read) ou l'analyse qPCR.
Notre stratégie de séquençage sera guidée par les caractéristiques de votre projet, chaque aspect, de l'extraction aux méthodes d'analyse, sera spécifiquement construit autour de ces caractéristiques.
 Méthodologie d'extraction
Méthodologie d'extraction
Utilisation de méthodes d'extraction à faible biomasse pour isoler et purifier l'ADN ou l'ARN à partir d'échantillons biologiques contenant une quantité limitée de matériel génétique.
Pipelines d'analyse propriétaires
Nous mettons à votre disposition des outils bioinformatiques et des bases de données pour l'analyse du microbiome avec des pipelines développés en interne et adaptés à la nature de l'échantillon étudié.

Interprétation des résultats
Vous recevrez des résultats clairs et compréhensibles, prêts à être publiés, ainsi qu'une présentation guidée pour vous assurer que vous tirez le meilleur parti de notre analyse.
 Rapport interactif
Rapport interactif
Quel que soit l'objectif de votre projet (recherche, marketing ou réglementation), les résultats seront adaptés à chacun.
Soutien d'experts
Un soutien scientifique, technique et commercial complet tout au long de votre projet, avec des conseils de microbiologistes de niveau doctoral.
 Partenaire dédié
Partenaire dédié
Vous travaillerez avec l'un de nos experts qui adaptera les solutions à vos besoins spécifiques.

Contactez-nous pour plus d'informations
Téléchargez notre rapport
Analyses du microbiome cutané
Analyse et caractérisation du microbiote de la peau
Le microbiote de la peau est constitué de milliards de micro-organismes, couvrant diverses espèces, vivant en communauté et étroitement liés à la santé de la peau. De plus en plus, cette microflore est étudiée à l'aide de solutions génomiques. Ces technologies ouvrent de nouvelles perspectives pour le développement de produits de soins cosmétiques et de traitements personnalisés pour les peaux atopiques. Grâce à son expérience et son savoir-faire, GenoScreen a ainsi développé une solution complète pour l'analyse du microbiome cutané.
Ce que la génomique apporte à notre compréhension du microbiome cutané
La métagénomique représente l'approche la plus complète et innovante pour caractériser et étudier les microbiomes, permettant d'avoir accès à l’ensemble des microorganismes présents dans tout échantillon, et à leur potentiel fonctionnel en identifiant des gènes d’intérêt impliqués dans des voies métaboliques ayant une influence sur la santé de la peau.
Dans ce cadre, GenoScreen intervient à différentes étapes de tout projet de recherche et développement sur le microbiome cutané. Nos solutions génomiques couvrent les objectifs suivants :
- Études d’impact sur le microbiote cutané d’un produit cosmétique ou d’un ingrédient actif.
- Études cliniques en dermatologie ou dermocosmétique pour évaluer l’effet de traitement sur les pathologies affectant la peau.
- Projets exploratoires en R&D axées sur le microbiome cutané
- Objectivation d’actifs et preuves de concept des effets de tels ingrédients sur les micro-organismes présents sur l’épiderme
- Support de la communication scientifique avec des résultats d’études prêts pour leur publication.
- Validation d’allégation scientifiques pour démontrer l’interaction d’un produit avec le microbiome de la peau et mieux comprendre son mode d’action.
- Certification d’allégations « microbiome friendly» et « microbiome tested», confirmant auprès des consommateurs l’innocuité d’un produit cosmétique pour la santé de la peau et l’intégrité de son microbiome.
- Tests d’ingrédients prébiotiques, probiotiques, postbiotiques
- Collaboration R&D pour des projets d’études exploratoires des communautés microbiennes : détection de biomarqueurs, analyses ciblées, par exemple.
Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser.
Nos solutions pour le test et l’analyse du microbiome cutané
De votre question biologique initiale à sa réponse technique, nous mettons en commun notre savoir-faire et nos technologies pour vous apporter la solution la plus adéquate pour répondre aux besoins de votre projet.
Principalement réalisées à partir d’échantillons cutanés prélevés sur de l’humain, dans le cadre d’études in vivo , nos analyses peuvent être conduites sur d’autres modèles : peau humaine de synthèse (RHE), explants et culturomique (échantillons in vitro ou ex vivo).
Métagénomique
La clé pour comprendre les interactions entre l’épiderme, sa santé et sa flore microbienne.
- Métagénomique ciblée « Short read » : La première étape d’analyse du microbiote cutané. Sur une plateforme de séquençage Illumina, la métagénomique ciblée en short read permet d’obtenir un aperçu global des genres taxonomiques des micro-organismes en présence dans les échantillons issus d’écouvillonnage sur la peau. L’identification de la microflore cutanée repose sur le séquençage de la région 16S de l’ADN ribosomique (rDNA) pour les cibles bactérienne ou sur la région 18S/ITS du rDNA pour les cibles fongiques.
- Métagénomique ciblée « Long read » : La solution la plus adaptée à l’analyse et la caractérisation du microbiome de la peau. La métagénomique long read permet d’aller plus loin, plus précisément dans l’identification taxonomique, au niveau de l’espèce. Un avantage considérable quand il est question de distinguer les différentes espèces d’importance pour la santé de la peau. Basée sur une plateforme PacBio ou Oxford Nanopore Technologies (ONT), la métagénomique en long read permet également une comparaison plus détaillée entre les échantillons.
- Métagénomique Shotgun : Description précise et complète de tous les génomes de micro-organismes dans chaque échantillon. La métagénomique Shotgun offre un potentiel fonctionnel du microbiome cutané, au-delà de la caractérisation de toutes les espèces en une seule analyse comme les procaryotes (bactéries, levures et autres champignons) et les petits eurcaryotes. Grâce au séquençage de nouvelle génération (NGS) sur une plateforme Illumina, cette approche non ciblée offre une vision globale des génomes de tous les organismes constituant le microbiote cutané et donne accès à une quantification en valeur relative de leur contenu et de leur proportion dans chaque échantillon. Avec cette technologie, l'annotation des gènes est permise.
-
- Prise en compte de tous les microorganismes en présence (tous domaines, règnes et genres confondus) en une seule analyse
- Quantification relative des populations au sein des échantillons
- Vue d’ensemble complète du génome du microbiote
- Annotation des gènes d’intérêt
- Accès au potentiel fonctionnel du microbiome de la peau
Bioinformatique
- Expertise technique
Notre plateforme de bioinformatique base son travail sur des bases de données et des pipelines spécifiques des microbiomes cutanés, développés en interne, et permettant une analyse en profondeur des communautés microbiennes.
- Décrypter le microbiome de la peau
Nous fournissons, à travers des rapports interactifs, un panorama complet du microbiote cutané intégrant, entre autres paramètres, les suivants : alpha-diversité et beta-diversité, distance entre les échantillons et comparaison de leur composition.
- Interface simple d’utilisation
L’interface est à la fois interactive pour l’utilisateur et d’autre part bâtie dans un format tel qu’il permet son utilisation pour des publications ou de la communication scientifiques, des dossiers réglementaires ou d’études cliniques, tout en rendant son contenu accessible et compréhensible pour l’utilisateur.
Approches qPCR
La conception et l’ingénierie d’amorces spécifiques à une espèce ou souche composant le microbiome cutané, et de sondes spécifiques pour leur quantification, ce dans divers types d’échantillons : in vivo , ex vivo , in vitro.
La PCR quantitative en temps réel permet une évaluation rapide, robuste et fiable des effets de l’applications de produits de soin de la peau sur le microbiote cutané.
GenoBiome Skin
Solution globale d’analyse du microbiome cutané, adaptable à chaque projet, à partir d’échantillons prélevés in vivo. Un large panel de solutions d’analyses génomiques bénéficiant de l’accompagnement et de l'expertise de notre équipe de microbiologistes et de bioinformaticiens. GenoBiome Skin est basé sur un pipeline complet, du design du protocole expérimental avec nos experts, en passant par le séquençage et le traitement des données en bioinformatique, jusqu'au rendu et à l'interprétation des résultats. Parmi la gamme de réponses technologiques pouvant s’appliquer dans le cadre de GenoBiome Skin, on retrouve notre portefeuille métagénomique ainsi que l'analyse personnalisée de souches spécifiques.
GenoBiome Skin constitue à ce jour la solution la plus avancée pour vous accompagner dans vos projets de recherche et de développement sur le microbiote de la peau.
- Design expérimental de tests in vivo
- Protocole standardisé d’échantillonnage cutané
- Fourniture de matériel de prélèvement (écouvillons, swabs)
- Protocole de conservation et d’expédition des échantillons
- Pipeline d’analyse sur mesure, avec des méthodes d’extractions pour les biomasses rares et une stratégie de séquençage optimisée
- Bases de données et outils bioinformatiques spécialisés pour analyser le microbiome cutané
- Rapports interactifs, prêts à être publiés
- Interprétation des résultats : plus que des données de séquençage, nous donnons des réponses à vos questions biologiques
- Accompagnement tout au long de votre projet, jalonnés par des rendez-vous de suivi réguliers

Intéressés par nos solutions pour votre projet ? Contactez-nous et communiquez-nous les paramètres de votre future étude : nombre d’échantillons, protocole initial (fréquence de prélèvements), panel sélectionné, objectif (question biologique), réponse recherchée et technologie associée (quel niveau d’analyse vous intéresse-t-il), nos équipes se chargeront de vous fournir une réponse rapide et adaptée.
Caractérisation et traçage des microorganismes isolés
A partir d’un panel complet de technologies et de services en génomique, GenoScreen propose une variété de solutions d’identification de microorganismes par séquençage d’ADN et de comparaison aux bases de données internationales. Puissantes et précises, ces solutions identifient et caractérisent très précisément les microorganismes d’intérêt : identification à l’espèce, à la sous-espèce ou à la souche, typage, traçage et antibiorésistance.
L’identification et la caractérisation
Pour une identification à l'espèce, les régions de l’ADN ribosomique interrogées sont adaptées à chaque micro-organisme.
En complément, d’autres cibles génomiques (gènes rpoB, gyrB…) sont utilisées pour affiner l’identification et l’adapter à votre demande (discrimination au sein d’un groupe d’espèces partageant la même séquence d’ADN ribosomique, par exemple).
GenoScreen peut aussi mobiliser ses savoir-faire en séquençage de génome complet (WGS) pour une caractérisation plus précise des souches : évaluation de la virulence et de la résistance aux antibiotiques, comparaisons génomiques sur le souchier…
Le typage et le traçage microbien
Notre solution caractérise précisément tout microorganisme à la sous-espèce ou à la souche à l’aide de méthodes moléculaires standardisées et validées de type MLST (MultiLocus Sequencing Typing) ou MLVA (Multiple Loci VNTR Analysis).
Les schémas de typage publics sont utilisés comme référence pour ces analyses (PubMLST, MLST.org et MicrobesGenotyping). L’analyse peut aussi être réalisée sur la base d’un schéma de typage personnalisé.
GenoScreen, leader mondial du génotypage de la tuberculose
Grâce à un savoir-faire technologique de pointe, GenoScreen est aujourd’huile leader mondial pour le génotypage et le traçage des souches du complexe Mycobacterium tuberculosis, agent responsable de la tuberculose.
La solution MIRU-VNTR développée par nos équipes est utilisée par de nombreux centres de soins et équipes de recherche sous forme de kit ou de prestation de service. L’approche complémentaire par spoligotypage est également possible, afin d'identifier des sous-espèces.
Nous proposons des kits et des formations pour l'implémentation et l'usage optimisé de la méthode MIRU-VNTR sur séquenceurs Applied Biosystems® et QIAGEN®, directement dans vos laboratoires hospitaliers ou de recherche.
L'antibiorésistance
GenoScreen réalise des antibiogrammes moléculaires par séquençage ciblé de gènes connus pour être associés à la résistance aux antibiotiques ou par séquençage de génome complet. Les séquences sont comparées aux banques de données publiques accessibles ou aux informations disponibles dans la littérature.
Le séquençage en profondeur NGS permet de détecter la présence de variants minoritaires pouvant conférer la résistance (hétérorésistance).
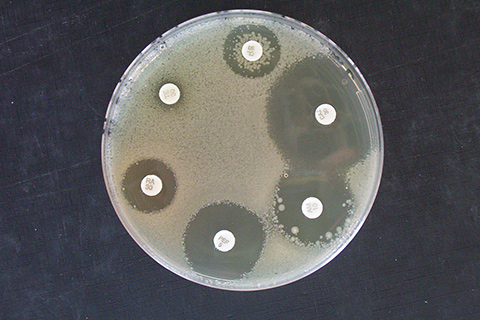
La solution Deeplex® Myc-TB développée par GenoScreen est une innovation majeure pour l’identification, la caractérisation et la surveillance épidémiologique de la tuberculose à des fins de recherche.
Deeplex® Myc-TB constitue un test tout-en-un permettant l’identification à l’espèce des mycobactéries, le génotypage et la prédiction de la résistance aux antibiotiques pour les souches du complexe Mycobacterium tuberculosis.
Accueil GenoScreen Expertise
La microbiologie décryptée
Leader international des services de séquençage et de génotypage, GenoScreen a développé deux expertises de pointe dans l’analyse génomique des agents microbiens et des communautés microbiennes.
A propos - Expertise
Une expertise dans le décryptage génomique des microorganismes
Spécialiste international de la génomique appliquée à la microbiologie, GenoScreen développe des solutions rapides et précises de caractérisation, de traçage et de typage de microorganismes isolés (bactéries, champignons, levures…) et d’analyse des flores microbiennes complexes :
- Services scientifiques et technologiques à haut niveau d’expertise,
- Travaux de recherche sous contrat,
- Formations spécialisées.
Ces stratégies d’analyses avancées répondent à de nombreuses problématiques de recherche dans les secteurs de la santé, la cosmétique, l’environnement, les agro-industries, la recherche académique, les biotech et les sols : elles identifient, tracent, décryptent et caractérisent les microorganismes, soit à l’échelle de l’individu soit à celle des communautés microbiennes, afin de comprendre leurs impacts positifs comme négatifs sur la santé, la sécurité et le bien-être des humains, des animaux et de l’environnement.
Exemples de projets développés
- Mise au point d’un processus de caractérisation génomique de bactéries génétiquement modifiées,
- Etude des effets d’une molécule visant à protéger le microbiote intestinal des effets des traitements antibiotiques,
- Mesure d’impact de probiotiques et autres compléments alimentaires sur le microbiote digestif de certains animaux d’élevage, dans le but d’accélérer leur développement,
- Etude du rétablissement de l’équilibre de la flore microbienne d’ulcères en cours de traitement,
- Etude de l’impact de crèmes de soin sur le microbiote de la peau (avec GenoBiome® Skin),
- Développement d’un outil d’analyse métatranscriptomique de flores microbiennes agissant dans des eaux de station d’épuration.


- Ingénieries de caractérisation, de typage et de génotypage de microorganismes,
- Ingénieries d’analyse structurelle et fonctionnelle de métagénomes,
- Bioinformatique et biostatistique.
Analyse de communautés microbiennes (microbiotes)
L’analyse des microbiotes permet d’étudier l’ensemble des microorganismes présents dans un échantillon donné : bactéries, levures, champignons, algues…
Cette analyse est aujourd’hui cruciale pour faire progresser la recherche dans les secteurs de la santé humaine et animale, de la nutrition, de l’environnement et des biotechnologies. Leader des services d’analyse génomique, GenoScreen propose des solutions d’analyse des microbiotes puissantes et complètes : un véritable accélérateur de recherche et une porte ouverte sur la génomique de demain !
Comprendre des écosystèmes complets, à l’échelle moléculaire
Les analyses moléculaires de communautés microbiennes ont surtout l’avantage, par rapport à la microbiologie classique, de permettre l’étude des microorganismes au sein même de leur niche écologique, sans devoir passer par des étapes de culture sur milieux synthétiques qui les isolent de leur communauté et de leur environnement naturel.
Ces analyses d’écologie microbienne détaillent la biomasse, la composition, la diversité, les fonctions et les interactions des micro-organismes entre eux et avec leur habitat.










