Français
Politique de confidentialité
Politique de Confidentialité des données
Veuillez lire attentivement la Politique de Confidentialité suivante (la « Politique de Confidentialité ») qui décrit comment GenoScreen (le « Responsable de Traitement ») recueille, utilise, divulgue lorsque nécessaire, stocke et gère de toute autre manière les informations personnelles identifiables (les « Données Personnelles »). La Politique de Confidentialité s'applique à tous les sites web, applications ou services gérés ou offerts par GenoScreen. GenoScreen peut modifier cette politique en publiant une version révisée sur le site et sans vous donner de préavis. Veuillez donc vérifier cette politique chaque fois que vous visitez le site et notez la date de publication. Lorsque vous nous fournissez des Données Personnelles de la manière décrite à l'article 2 de la présente Politique de Confidentialité, vous nous autorisez à les collecter, à les stocker et à les utiliser pour :
- Remplir nos obligations contractuelles et commerciales envers vous ;
- Administrer, soutenir, améliorer et développer notre entreprise d'une manière légalement justifiée (comme expliqué dans l'article 3 de cette Politique de Confidentialité).
Ceci étant fait avec votre consentement, vous êtes autorisé à le retirer à tout moment comme décrit dans l'article 6 de la présente Politique de Confidentialité.Le règlement européen sur la protection des données (RGPD) remplace la directive 95/46/CE sur la protection des données et vise à harmoniser les lois sur la confidentialité des données à travers l'Europe. Les articles clés du RGPD, ainsi que des informations sur son impact commercial, peuvent être consultés sur ce site: https://www.eugdpr.org/
Article 1 – Le Responsable de Traitement
Le Responsable de Traitement des Données Personnelles est GenoScreen, une société française dont le siège social est situé au 1 rue du Professeur Calmette - 59000 Lille, FRANCE, inscrite au registre du commerce de Lille Métropole sous le numéro 433996220.
Article 2 – La collecte de données
Sous réserve des lois applicables, nous recueillons des informations à votre sujet ou sur des tiers dont vous nous fournissez les informations lorsque vous :
- Vous inscrivez pour utiliser nos sites Web, applications ou services (y compris les essais gratuits) ; cela peut inclure votre nom (ou le nom de votre entreprise, le cas échéant), votre adresse, votre adresse électronique et votre numéro de téléphone. Nous pouvons également vous demander de nous fournir des informations supplémentaires sur votre entreprise et vos préférences ;
- Passez une commande en utilisant nos sites Web, applications ou services; cela peut inclure votre nom (ou votre raison sociale), votre adresse et vos coordonnées (y compris votre numéro de téléphone et votre adresse électronique) et votre paiement ;
- Remplissez des formulaires en ligne (y compris des demandes de rappel), répondez à des sondages, téléchargez des informations telles que des livres blancs ou d'autres publications. espaces présents sur notre site web ou sur notre application ou service ;
- Interagissez avec nous en utilisant les réseaux sociaux ;
- Fournissez vos coordonnées lorsque vous vous inscrivez ou accédez à un site Web, une application ou un service que nous mettons à votre disposition ou lorsque vous mettez à jour ces informations ;
- Lorsque vous nous contactez hors ligne, par exemple par téléphone, fax, SMS, e-mail ou courrier.
- Nous recueillons également vos Données Personnelles lorsque vous ne remplissez qu'une partie des champs à remplir sur notre site et/ou sur d'autres formulaires en ligne et/ou lorsque vous abandonnez cette entrée en cours de route. Nous pouvons utiliser ces données pour vous contacter afin de vous rappeler d'entrer des informations manquantes et/ou à des fins de marketing. Nous pouvons également collecter des informations de vos appareils électroniques (y compris les téléphones mobiles) et des applications utilisées par vous ou vos utilisateurs pour accéder et utiliser nos sites web, applications ou services (par exemple, nous pouvons collecter le numéro d'identification et le type d'appareil utilisé, géolocalisation et informations de connexion, telles que les statistiques sur les pages visitées, le trafic depuis et vers les sites, l'URL de référence du lien, les données d'annonce, votre adresse IP, votre historique de navigation et vos informations de connexion web). Nous vous demanderons votre autorisation à l'avance avant toute étape. Sous réserve des lois applicables, nous pouvons compléter les Données Personnelles que nous collectons avec des informations obtenues par des tiers autorisés à les partager ; par exemple, des informations émanant d'établissements de crédit, de fournisseurs à la recherche d'informations ou de sources publiques (par exemple, dans le cadre de la vigilance à l'égard de la clientèle).
Données de tiers
Si vous nous fournissez des Données Personnelles concernant un tiers, il est de votre responsabilité de vous assurer que vous respectez les réglementations applicables en matière de protection des Données Personnelles et en particulier vos obligations d'obtenir le consentement préalable des personnes dont vous nous fournissez les Données Personnelles. En conséquence, conformément à la réglementation en vigueur sur la protection des données, vous devez avoir informé la personne concernée et obtenu son consentement exprès à nous communiquer ses Données Personnelles, ainsi que l’avoir l'informée de la manière dont nous collectons, utilisons, divulguons et conservons des données à son sujet, ou invitée à lire notre Politique de Confidentialité.
Article 3 – L’utilisation des données collectées
Sous réserve des lois applicables, nous collectons et traitons vos Données Personnelles aux fins suivantes :
- Fournir les informations et services que vous avez demandés et les applications ou services que vous avez commandés ;
- Comparer les informations dans le but de vérifier leur exactitude et de les corroborer avec celles que possèdent des tiers ;
- Fournir, maintenir, protéger et améliorer les applications, produits, services et informations que vous nous avez demandés ;
- Gérer et administrer l'utilisation que vous faites des applications, produits et services que vous nous avez demandé de fournir ;
- Gérer notre relation d'affaires (par exemple, les services à la clientèle et les activités de soutien) ;
- Surveiller, mesurer, améliorer et protéger notre contenu, nos sites web, nos applications et nos services et vous fournir une expérience utilisateur personnalisée et de qualité ;
- Effectuer, en interne, des contrôles sur nos sites web, applications, systèmes et services afin de tester et d'améliorer leur sécurité, leur mise à disposition et leurs performances. Le cas échéant, nous utiliserons une forme pseudonymisée de toute information utilisée à ces fins, et nous veillerons à ce que ces informations soient présentées dans un emballage et ne soient pas contraignantes pour vous ou toute autre personne impliquée ;
- Vous fournir toute information que nous sommes tenus de vous fournir afin de nous conformer à nos obligations réglementaires ou légales ;
- Détecter, prévenir, enquêter ou empêcher des activités criminelles, illégales ou interdites, ou protéger nos droits (y compris la liaison avec les organismes d'application de la loi et de réglementation à ces fins) ;
- Vous contacter pour savoir si vous souhaitez participer à nos enquêtes clients (par exemple, des commentaires sur votre utilisation de nos applications, produits et services) ;
- Suivre et analyser conjointement les statistiques et les comparaisons pour éviter d'être identifié ou d'identifier toute autre personne ;
- Vous fournir des publicités, des messages marketing ou des informations ciblées qui pourraient vous être utiles, en fonction de votre utilisation de nos applications et services ;
- Livrer du contenu et des services conjointement avec des tiers avec lesquels vous avez une relation distincte.
- Vérifier les informations spécifiques de votre appareil ou des systèmes nécessaires à votre utilisation des sites web, applications ou services en relation avec nos enregistrements pour garantir que les sites web, applications ou services sont utilisés conformément à nos conditions générales et nos accords avec l'utilisateur final et dans le but de résoudre des problèmes potentiels ;
- Obtenir des informations sur des erreurs techniques ou d'autres problèmes liés à nos sites web, applications et services ;
- Recueillir des informations sur la manière dont vous et les utilisateurs utilisez les fonctionnalités de nos sites web, applications et services.
Vous pouvez gérer vos paramètres de confidentialité dans votre navigateur ou sur nos applications ou services (le cas échéant).
GenoScreen utilise Sarbacane
GenoScreen envoie des communications par e-mail notamment via un tiers nommé Sarbacane. Plus de 12 millions de personnes et d'entreprises à travers le monde utilisent Sarbacane. Les fonctionnalités et intégrations Sarbacane permettent à leurs clients d'envoyer des e-mails marketing, des messages automatisés et des campagnes ciblées.
Article 4 – Le partage de données
Nous sommes susceptibles de partager vos Données Personnelles avec :
- Nos fournisseurs de services et agents (y compris leurs sous-traitants) ou les tiers qui traitent les informations en notre nom (par exemple, plateformes Internet ou fournisseurs de services, processeurs de paiement et sociétés que nous utilisons pour nous aider à vous envoyer des communications) vous avec les applications, produits, services et informations que vous avez été intéressé à recevoir ou qui, de notre point de vue, seraient susceptibles de vous intéresser ;
- Des partenaires, notamment des constructeurs de systèmes, des revendeurs, des distributeurs, des éditeurs de logiciels et des développeurs, qui nous aident à vous fournir les applications, produits, services et informations que vous avez demandés ou qui sont susceptibles de vous intéresser ;
- Les tiers auxquels nous recourons pour l'exécution des opérations de paiement, tels que les sociétés de compensation, les systèmes de compensation, les institutions financières et les bénéficiaires de transactions ;
- Les tiers, lorsque vous entretenez une relation avec ce tiers et que vous avez consenti à ce que nous transmettions des informations (par exemple, des sites de réseaux sociaux ou d'autres fournisseurs d'applications tiers) ;
- Les tiers à des fins de marketing (par exemple nos partenaires et autres tiers avec lesquels nous travaillons et dont les produits, de notre point de vue, peuvent vous intéresser dans la conduite de vos activités commerciales, par exemple : les fournisseurs de services financiers (tels que en tant que banques, assureurs et fournisseurs de services financiers), fournisseurs de solutions de paiement, fournisseurs de logiciels et fournisseurs de services qui fournissent des solutions commerciales) ;
- Les agences de référence de crédit et de prévention de la fraude ;
- Les régulateurs, pour respecter les obligations légales et réglementaires de GenoScreen ;
- Les autorités policières, afin qu'elles puissent détecter ou prévenir les crimes ou poursuivre les contrevenants ;
- Tout tiers, dans le cadre de procédures judiciaires existantes ou en cours, à condition que nous ayons légalement le droit de le faire (par exemple, en réponse à une ordonnance rendue par un tribunal) ;
- Toute tierce partie à se conformer à nos obligations légales et réglementaires, y compris les rapports légaux ou réglementaires et la détection ou la prévention d'actes illicites ;
- Nos propres auditeurs et consultants, pour remplir nos responsabilités d'audit ;
- Une autre société, si nous vendons ou achetons une activité ou des actifs (ou négocions la vente ou l'achat d'une activité ou d'actifs) ;
- Toute autre société à laquelle nous pourrions être tenus de céder le contrat qui nous lie ;
- Les organismes publics que la loi en vigueur exige d'informer.
Nous sommes susceptibles de partager, publiquement ou avec des tiers, des informations sur nos sites web, applications, produits ou services à l'exclusion de toute information pouvant vous identifier.
Article 5 – Marketing
Nous pouvons parfois utiliser vos informations pour vous contacter dans le but de vous fournir des informations sur nos applications, produits et services susceptibles de vous intéresser. À cette fin, vous pouvez être contacté par téléphone, courrier, SMS ou e-mail. Vous avez le droit de nous demander d'arrêter de vous contacter à des fins de marketing à tout moment. Si vous le souhaitez, vous pouvez exercer ces droits en sélectionnant vos préférences de contact à l'endroit où vous nous fournissez vos Données Personnelles sur nos sites, applications ou services, en utilisant n'importe quel centre de préférences auquel nous vous donnons accès ou en nous, en envoyant un e-mail à Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser.. Vous pouvez également vous désabonner de toute campagne marketing et/ou publicité par e-mail en cliquant sur le lien inclus dans les emails que nous vous envoyons.
Article 6 – Vos droits
Vous aurez dans certains cas les droits suivants (pour plus de détails, voir https://www.eugdpr.org/) :
- Le droit de savoir comment nous utilisons vos données et le droit d'accès à vos Données Personnelles ;
- Le droit de demander la modification ou la suppression de vos données ainsi que de limiter le traitement de vos données ;
- Le droit de s'opposer au traitement de vos données, par exemple à des fins de marketing ou lorsque leur utilisation est basée sur notre intérêt légitime ;
- Le droit de recevoir des données à votre sujet que vous avez fournies automatiquement dans un format structuré, communément utilisé et lisible par machine, ou de les faire envoyer directement à une autre entreprise, si cela est techniquement faisable (« Portabilité des Données ») ;
- Lorsque vous avez consenti au traitement de vos données, le droit de retirer votre consentement conformément aux limitations légales ou contractuelles ;
- Le droit de refuser toute décision basée sur le traitement automatisé de vos Données Personnelles, y compris le profilage ;
- Le droit de déposer une plainte auprès de l'autorité de surveillance chargée des questions de protection des données (par exemple, la Commission Nationale de l’Informatique et des Libertés).
Si vous demandez à recevoir une copie de vos données, vous devrez peut-être payer certains frais. Si nous avons des informations incorrectes à votre sujet ou si vos coordonnées ont changé, nous vous remercions de nous en informer afin que nous puissions corriger et mettre à jour nos fichiers. Si vous retirez votre consentement à l'utilisation de vos Données Personnelles aux fins énoncées dans notre Politique de Confidentialité, nous pourrions, par conséquent, ne plus être en mesure de vous fournir l'accès à tout ou partie de nos sites web, applications et services. Nous conserverons vos Données Personnelles pour la durée de notre relation d'affaires et après son expiration pour la période de temps nécessaire pour la conduite légitime de nos affaires, ou pour se conformer aux lois et règlements applicables. Dans le cas où nous n'aurions plus besoin de vos Données Personnelles, nous les détruirons de manière sécurisée (sans vous envoyer d'autre avis).
Article 7 – Stockage et sécurité des données
Nous assurons la sécurité de vos données en prenant les mesures techniques et structurelles nécessaires pour empêcher leur traitement illégal ou non autorisé ou la perte accidentelle, la destruction et/ou les dommages. Nous nous efforçons de protéger vos Données Personnelles autant que nous le pouvons. Cependant, nous ne pouvons pas garantir la sécurité de vos données transmises à nos sites Internet, applications ou services ou à d'autres sites Internet, applications et services via une connexion Internet ou toute autre connexion. Si nous vous avons donné (ou si vous avez choisi) un mot de passe vous permettant d'accéder à certaines zones de nos sites web, applications ou services, veuillez le garder confidentiel. Nous ne partagerons ce mot de passe avec personne. Si vous pensez que votre compte a été piraté, veuillez nous contacter à l'adresse dpo@genoscreen.com.
Article 8 – Données transférées hors de l’EEE
Dans l'Union européenne, les Données Personnelles sont protégées par des lois sur la protection des données. Cependant, d'autres pays ne protègent pas nécessairement vos Données Personnelles de la même manière. Nos sites web, nos services et applications peuvent également être hébergés aux États-Unis ou en dehors de l'EEE (qui est composé de l''Union européenne ainsi que la Norvège, l'Islande et le Liechtenstein). Cela implique que nous pouvons transférer les données qui nous sont transmises via le site web, l'application ou le service en dehors de l'Espace économique européen (« EEE ») vers les États-Unis ou le Canada ou autres territoires en dehors de l'EEE. Nous pouvons utiliser les services de fournisseurs situés en dehors de l'EEE pour nous aider à vous fournir nos sites web, applications et services (par exemple, plateforme ou fournisseurs de services de paiement qui nous aident à fournir nos services et applications ou à effectuer vos paiements). Cela signifie que nous sommes susceptibles de transmettre vos données à des prestataires de services situés en dehors de l'EEE dans le but de vous fournir nos applications et services. Nous nous assurons que lorsque nos fournisseurs de services et nos hébergeurs transfèrent vos données à l'extérieur de l'EEE, des mesures et des contrôles appropriés sont mis en œuvre conformément aux lois et réglementations applicables pour les protéger. Dans chaque cas, ces transferts sont effectués conformément aux exigences du règlement (UE) 2016/679 (règlement général sur la protection des données) et peuvent être basés sur l'utilisation de clauses standard sur les transferts de données à caractère personnel en dehors de l'EEE de la Commission européenne. En utilisant nos sites Web, produits et services ou interagissant avec nous dans les conditions décrites dans cette Politique de Confidentialité, vous consentez au transfert de vos données hors de l'EEE dans les cas prévus dans cette Politique de Confidentialité. Si vous refusez de transférer vos données en dehors de l'EEE, vous ne pourrez pas utiliser nos sites Web, applications ou services.
Article 9 – Google Analytics
Google Analytics utilise des «cookies» pour permettre aux sites web d'analyser la manière dont les utilisateurs utilisent les sites web, les applications et les services. Les informations sur votre utilisation des sites Internet, des applications ou des services (y compris votre adresse IP) générés par le cookie sont transmises à Google pour stockage sur des serveurs situés aux États-Unis. Google utilisera ces informations pour évaluer votre utilisation des sites web, des applications et des services, pour compiler des rapports sur l'activité du site pour les opérateurs de site et pour fournir d'autres services liés à l'activité du site Web et l'utilisation d'Internet. Google peut également divulguer ces informations à des tiers lorsque la loi l'exige ou lorsque ces tiers traitent ces informations pour le compte de Google. Google n'associera pas votre adresse IP à d'autres données détenues par Google. Vous pouvez refuser l'utilisation de cookies en sélectionnant les paramètres appropriés dans votre navigateur ou votre application. Notez cependant que votre refus peut entraîner une indisponibilité partielle du site. Pour plus d'informations, reportez-vous à "Utilisation de certaines données recueillies par Google lorsque vous utilisez des sites ou des applications partenaires" (disponible sur www.google.com/policies/privacy/partners/ ou tout autre lien que Google peut vous fournir). Si vous souhaitez que Google Analytics cesse de vous suivre sur tous les sites web, consultez la page http://tools.google.com/dlpage/gaoptout.
Article 10 – Questions et commentaires
Si vous avez des questions ou des commentaires sur cette Politique de Confidentialité, envoyez-nous un email à Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser. ou écrivez à GenoScreen, 1 Rue du Professeur Calmette, 59000 Lille - FRANCE.
Mentions légales
Renseignements éditoriaux
Vous êtes connecté au site officiel de GenoScreen, 1 rue du Professeur Calmette, 59000 Lille, FRANCE.
SIRET 433 996 220 00029
Société immatriculée au RCS de Lille
N° TVA intra. FR 61 433 996 220
SAS au capital de 463 032€
Directeur de la publication : André TORDEUX, Président - Directeur Général
Responsable éditorial : Raphaëlle POUJOL, Chargée de communication
Conception graphique et développement, ressources visuelles : Staminic
Pour toute remarque sur le contenu ou le fonctionnement du site, contactez notre webmaster;
Droits de reproduction
La mise en place de liens vers le site www.genoscreen.com doit avoir obtenu l'autorisation du responsable éditorial. Les pages de www.genoscreen.com ne doivent pas être imbriquées à l'intérieur des pages d'un autre site. La reproduction du contenu ou de pages du site requiert l’autorisation préalable du responsable éditorial et de mentionner le copyright de GenoScreen.
Notes
Copyright GenoScreen.
GenoScreen vérifie et met à jour toutes les informations contenues dans ce site. Néanmoins, toutes les informations peuvent être sujettes à modification et devenir obsolètes. Par conséquent GenoScreen ne peut assumer aucune garantie pour l'actualité, l'exactitude et l'exhaustivité des informations fournies.
Conformément à la loi Informatique et Libertés, vous disposez d'un droit d'accès, de rectification et de suppression des données vous concernant. Pour l'exercer, contactez GenoScreen.
Politique de confidentialité
Pour consulter notre Politique de confidentialité, cliquez ici.
Conditions générales de vente
Pour consulter et télécharger nos conditions générales de vente, cliquez ici.
Si vous avez des questions ou des commentaires sur cette Politique de Confidentialité,
envoyez-nous un email à Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser. ou écrivez à GenoScreen, 1 Rue du Professeur Calmette, 59000 Lille - FRANCE.
Qualité et certifications - Exigence et excellence au service de nos clients
GenoScreen a fait très tôt de la qualité une priorité. Ce choix garantit à nos clients et à nos partenaires une organisation et des processus qui répondent parfaitement à leurs exigences. Elle permet aussi d’assurer le respect total de l’ensemble des réglementations applicables à ses services, y compris au niveau international.
La Direction de GenoScreen s’engage à garantir de façon permanente :
- La qualité et la fiabilité des prestations qu’elle réalise,
- La performance et la fiabilité des produits qu’elle propose,
- La sincérité des résultats qu’elle communique,
- La satisfaction durable de ses clients,
- L’adaptation de ses services aux attentes du client,
- Une amélioration continue de son organisation.
L’engagement de qualité traduit aussi les valeurs qui structurent notre action :
- Recherche de la satisfaction des clients,
- Amélioration continue de la qualité des services et des produits proposés,
- Recherche de la performance par l'innovation.
Certification ISO 13485 : 2016, Dispositifs médicaux - Systèmes de management de la qualité - Exigences à des fins réglementaires
GenoScreen est certifiée à la norme ISO13485 pour la « conception, développement, fabrication, commercialisation et distribution de kits de diagnostic in vitro. ». Cette certification complète la certification ISO9001.
Certification ISO 9001 : 2015, Système de management de la qualité - Exigences
 GenoScreen est certifiée à la norme ISO9001 pour la « Conception, Développement, Production et Commercialisation de l’ensemble des produits, prestations, expertises et formations en génomique et en bioinformatique ».
GenoScreen est certifiée à la norme ISO9001 pour la « Conception, Développement, Production et Commercialisation de l’ensemble des produits, prestations, expertises et formations en génomique et en bioinformatique ».
Autorisation MOT (Micro-organismes et toxines hautement pathogènes)
 GenoScreen est titulaire d’une autorisation de l’Agence nationale de sécurité du médicament et des produits de santé (ANSM) pour la détention, la mise en œuvre et l’emploi de micro-organismes et toxines hautement pathogènes. Cette autorisation garantit la mise en œuvre sécurisée des MOT et la traçabilité complète des échanges de MOT.
GenoScreen est titulaire d’une autorisation de l’Agence nationale de sécurité du médicament et des produits de santé (ANSM) pour la détention, la mise en œuvre et l’emploi de micro-organismes et toxines hautement pathogènes. Cette autorisation garantit la mise en œuvre sécurisée des MOT et la traçabilité complète des échanges de MOT.
Agrément MOT (Micro-organismes et toxines hautement pathogènes) délivré par l’ANSM (validité 21/03/2030) : Autorisation de Détention, d’Acquisition, de mise en œuvre et d’emploi de MOT selon le décret n°2010-736 du 30 juin 2010 relatif aux micro-organismes et toxines.
GCLP : Good Clinical laboratory Practice
 Les bonnes pratiques de laboratoire clinique (GCLP : Good Clinical laboratory Practice) constituent un système de qualité international bien établi pour les laboratoires qui analysent des échantillons d'essais cliniques conformément aux réglementations mondiales en matière de bonnes pratiques cliniques (GCP : Good Clinical Practice).
Les bonnes pratiques de laboratoire clinique (GCLP : Good Clinical laboratory Practice) constituent un système de qualité international bien établi pour les laboratoires qui analysent des échantillons d'essais cliniques conformément aux réglementations mondiales en matière de bonnes pratiques cliniques (GCP : Good Clinical Practice).
Les exigences de ce système qualité garantissent les droits des patients, préservent leur confidentialité et assurent la fiabilité, la qualité et l'intégrité du travail et des résultats produits.
CIR : 2020-2024, Crédit Impôt Recherche
 GenoScreen bénéficie du Crédit Impôt Recherche, mesure du soutien aux activités de recherches et développement (R&D) des entreprises.
GenoScreen bénéficie du Crédit Impôt Recherche, mesure du soutien aux activités de recherches et développement (R&D) des entreprises.
Oxford Nanopore Technologies, Certified Service Provider
GenoScreen est un prestataire de services certifié par Oxford Nanopore Technologies, garantissant les plus hauts standards de qualité pour les projets de séquençage long-read sur GridION.
International - Un leader de la génomique
Leader sur de nombreuses technologies en génomique, GenoScreen s’est rapidement développée à l’international pour apporter ses solutions aux équipes de recherche du monde entier.
Un leader international
La société a des débouchés dans près de 40 pays.
Répartition du CA
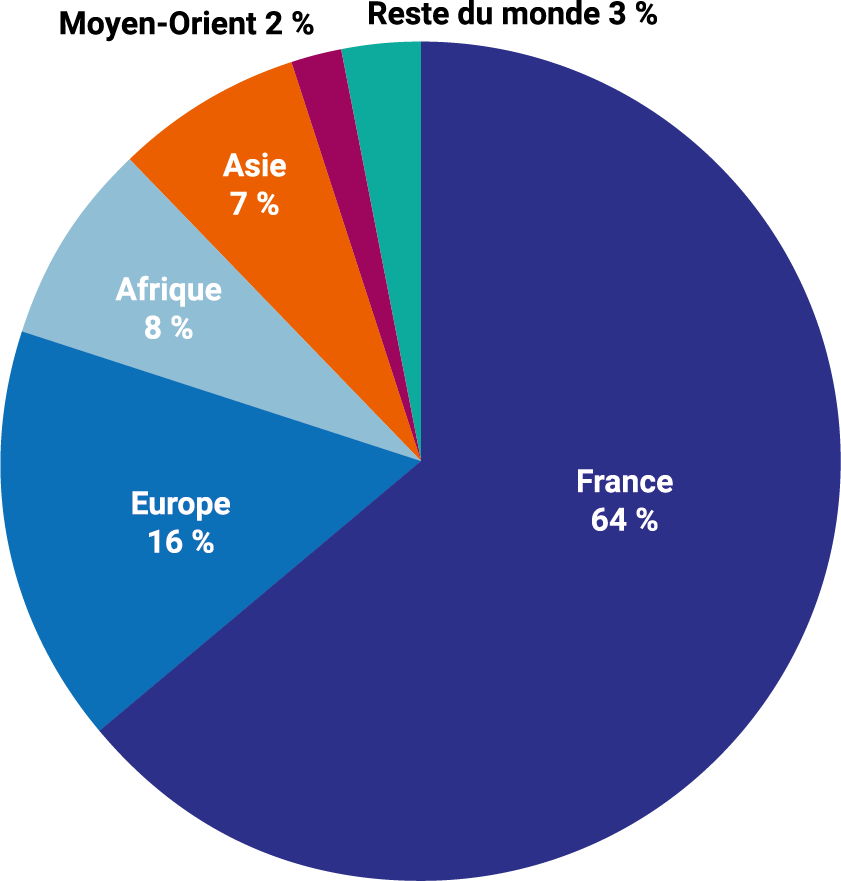
Des innovations de rang mondial
Des produits et des solutions, développés par GenoScreen, sont aujourd'hui des références internationales :
- Deeplex® Myc-TB, solution tout-en-un pour la caractérisation de Mycobacterium tuberculosis et la détection de ses antibiorésistances,
- Deeplex® Myc-Lep, solution pour la caractérisation de Mycobacterium leprae et la détection de ses antibiorésistances,
- GenoBiome®, notre offre intégrale pour l’analyse des communautés de microorganismes (de la peau avec GenoBiome® Skin).
Un acteur majeur dans les programmes de recherche internationaux
Les équipes de GenoScreen participent régulièrement à des programmes de recherche internationaux. Ces travaux lui permettent d’entretenir des relations étroites avec des équipes de recherche scientifique internationalement reconnues dans les domaines de la santé, de l’environnement, de la nutrition…
Sous-catégories
GenoScreen
Partenaires
GenoScreen développe ses technologies et savoir-faire en génomique au sein d’un réseau de partenaires dense et structuré.
Ces partenariats permettent de cofinancer des programmes de recherche ambitieux ou de co-développer des technologies innovantes : une stratégie efficace et agile, qui ouvre de larges possibilités à nos équipes.
Recherche collaborative - Au cœur de l’innovation génomique internationale
GenoScreen est partenaire de nombreux projets de recherche collaborative réunissant startups, grandes entreprises et organismes publics. Nos équipes de R&D y apportent leur savoir-faire et expertises de la microbiologie moléculaire d’agents isolés ou de communautés complexes. Nos équipes sont aussi à l’origine de projets visant l’élaboration de produits et services innovants basés sur l’utilisation et la connaissance des informations génomiques des microorganismes.
Nos interventions visent :
- L’amélioration de la détection et de la prise en charge thérapeutique de pathologies aigües ou chroniques chez l’homme ou l’animal,
- Le contrôle, la caractérisation et le suivi de la biodiversité microbienne, avec des applications dans l’agronomie, l’agroalimentaire et la protection de l’environnement.
Les objectifs communs à tous ces projets sont de développer des outils moléculaires pour la caractérisation, le suivi et le diagnostic des communautés microbiennes avec l’objectif ultime de mettre sur le marché des solutions analytiques simples mais aussi à terme des produits préventifs/correctifs ou même thérapeutiques basés sur les microorganismes.









